Tổng hợp và biên dịch: Nguyễn Huỳnh Thanh Tuấn
Khoa Y – Đại học Quốc gia Thành phố Hồ Chí Minh
Email: tuannguyenhuynhthanh@gmail.com
Tiếp theo phần trước.
Sử dụng công nghệ mới để tạo dòng muỗi “kháng sốt rét”
Không chỉ dừng lại ở đó, trong những năm trở lại đây, sự phát triển của khoa học – kỹ thuật đã giúp cho chúng ta có thêm những công cụ hết sức mạnh mẽ và hiệu quả trong việc chỉnh sửa cũng như cải biến vật chất di truyền ở cấp độ tận cùng – DNA hay bộ gene. Trong số đó, không thể không nhắc đến hệ thống CRISPR/Cas9. Hệ thống này dựa trên cơ chế “miễn dịch” của vi khuẩn chống lại sự xâm nhập của DNA ngoại lai có nguồn gốc từ virus hoặc DNA plasmid (10). Mặc dù tương đồng với enzyme cắt giới hạn về chức năng, song CRISPR/Cas9 lại có những khác biệt rất rõ rệt về cơ chế hoạt động. Hệ thống này dựa trên phân tử RNA để nhận diện và phá hủy DNA ngoại lai. Để có thể làm được điều này, vi khuẩn đã chèn một đoạn ngắn DNA ngoại lai vào bộ gene của mình tại vùng trình tự lặp lại CRISPR. Sau đó, vùng trình tự này được phiên mã và được xử lý thành các đoạn RNA ngắn (được gọi là crRNA). Tiếp đến, các crRNA này sẽ liên kết với endonuclease Cas9 để nhận diện DNA mục tiêu thông qua liên kết bổ sung giữa trình tự crRNA và DNA mục tiêu. Cuối cùng, DNA mục tiêu sẽ bị cắt thông qua hoạt tính endonuclease của Cas9.
Nhận thấy ưu điểm vượt trội của hệ thống này, các nhà khoa học đã tiếp nhận và biến đổi CRISPR/Cas9 từ một cơ chế “miễn dịch” của vi khuẩn thành một công nghệ biến đổi gene được đánh giá là có tiềm năng ứng dụng vô cùng mạnh mẽ. Trong lĩnh vực nghiên cứu về sốt rét, vào năm 2015, một số nhà khoa học đã sử dụng công nghệ này để tạo ra những con muỗi có những kiểu hình khác thường nhằm phục vụ cho việc nghiên cứu về đặc tính sinh học của loài này (11).
Và gần đây nhất, vào ngày 08/03/2018, các nhà khoa học đến từ Đại học John Hopkins đã công bố nghiên cứu của mình trên tạp chí Plos Pathogens (12). Theo đó, họ đã sử dụng hệ thống CRISPR/Cas9 để làm bất hoạt gene mã hóa cho Fibrinogen-related protein 1 (FREP1) ở muỗi Anopheles gambiae (A. gambiae). Kết quả thu được cho thấy những con muỗi bị bất hoạt gene này có khả năng kháng được ký sinh trùng sốt rét P. falciparum.
Như đã trình bày, các chất chủ vận và thụ quan đã trở thành những đối tượng trung tâm cho việc nghiên cứu các phương thức mới để kháng lại sốt rét và tiến tới kiểm soát dịch bệnh này một cách có hiệu quả, trong bối cảnh mà “kháng kháng sinh” đã trở thành một vấn đề đáng lo ngại. Thế nhưng, giữa hằng hà sa số các chất chủ vận cũng như thụ quan tương tác với nhau trong quá trình phát triển của ký sinh trùng sốt rét thì việc xác định được chất chủ vận hay thụ quan nào đóng vai trò then chốt, còn cái nào đóng vai trò thứ yếu không chỉ trong việc hỗ trợ mà còn cả ức chế sự phát triển của ký sinh trùng này là một thử thách rất lớn đối với các nhà khoa học. May mắn thay, những nghiên cứu trước đã cho thấy FREP1, một thụ quan ở chất nền peritrophic (peritrophic matrix) của thành dạ dày, đóng vai trò rất quan trọng trong việc hỗ trợ quá trình di chuyển của trứng “di động” (ookinete) qua thành dạ dày của muỗi để bước vào giai đoạn hình thành nang trứng (oocyst). (13, 14)
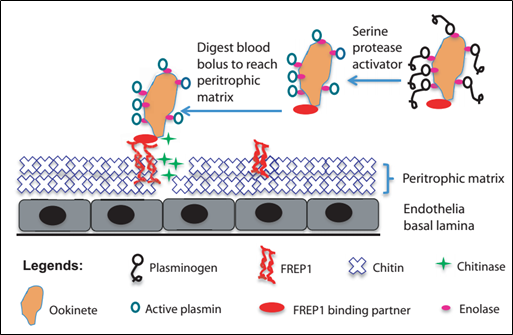
Hình 2. Mô hình về cơ chế phân tử điều hòa quá trình di chuyển của trứng “di động” qua thành dạ dày của muỗi nhờ vào FREP1 (14)
Quay trở lại với nghiên cứu của Đại học John Hopkins, để tạo dòng muỗi A. gambiae bị bất hoạt gen FREP1, các nhà khoa học đã tiến hành cho lai giữa dòng muỗi biểu hiện trình tự RNA nhận diện đối với gene FREP1 (FREP1 – gRNA) với dòng muỗi biểu hiện protein Cas9 ( Vasa – Cas9). Để đánh giá và so sánh mức độ lây nhiễm bởi ký sinh trùng Plasmodium, trước tiên họ phân loại thành 2 nhóm: nhóm thứ nhất là dòng muỗi có gene FREP1 bị bất hoạt (FREP1 – KOs) được tạo ra nhờ hệ thống CRISPR/Cas9; nhóm thứ hai gồm các dòng muỗi kiểu dại (wild type – X1), dòng muỗi FREP1 – gRNA và dòng muỗi Vasa – Cas9 để đối chứng (control). Tiếp đến, nhóm nghiên cứu cho các quần thể muỗi này hút máu có nhiễm các giao bào của ký sinh trùng P. falciparum lần lượt ở nồng độ cao (0.1%) và thấp (0.01%). Các kết quả được ghi nhận lần lượt sau 8 ngày và 14 ngày theo dõi không khỏi khiến ta bất ngờ:
Ở máu nhiễm giao bào của ký sinh trùng P. falciparum với nồng độ cao: Có đến 98,1% số muỗi trong quần thể kiểu dại có sự hiện diện của nang trứng tại mặt ngoài dạ dày. Không chỉ vậy, mật độ số nang trứng cũng cao bất thường (khoảng 91 nang trứng/cá thể muỗi). Trong khi đó, ở quần thể muỗi bị bất hoạt gene FREP1, tỷ lệ này thấp hơn một cách đáng kể khi chỉ rơi vào khoảng 75% và mật độ số nang trứng chỉ ở mức khoảng 17 nang trứng/ muỗi.
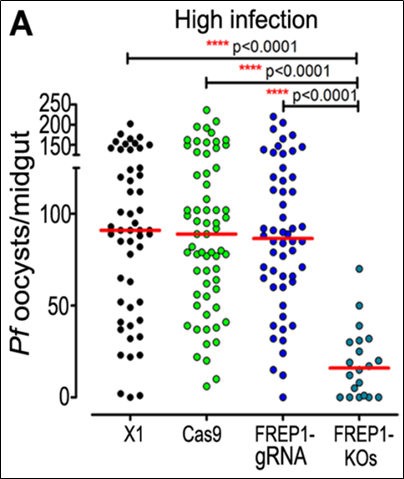
Hình 3. Biểu đồ định lượng mật độ số nang trứng hình thành ở mặt ngoài dạ dày khi cho các dòng muỗi hút máu có nhiễm giao bào của ký sinh trùng P. falciparum với nồng độ cao (12)
Ở máu nhiễm giao bào ký sinh trùng P. falciparum với nồng độ thấp: Trước tiên, cần phải nhấn mạnh rằng việc giảm đi nồng độ của giao bào ký sinh trùng này đi 10 lần nhằm mục đích để mô phỏng với điều kiện lây nhiễm ngoài tự nhiên (mimic a natural infection system). Vì vậy, các kết quả này sẽ cho giúp cho các nhà khoa học có được những hiểu biết gần với thực tế nhất. Theo đó, việc gây bất hoạt gene FREP1 ở muỗi gây ức chế rất mạnh đối với sự phát triển của giao bào ký sinh trùng P. falciparum khi mà đa số cá thể của dòng muỗi này không cho thấy bất kỳ sự hiện diện nào của các nang trứng được hình thành ở mặt ngoài dạ dày, cũng như số thoa trùng (sporozoites) đếm được ở tuyến nước bọt của dòng muỗi bị bất hoạt gene FREP1 sau ngày thứ 14 là cực kỳ thấp.
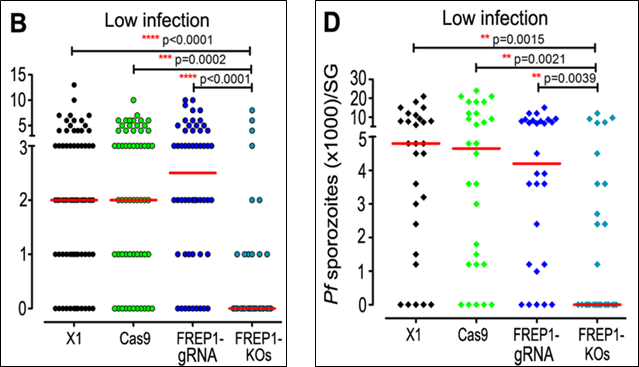
Hình 4. Biểu đồ định lượng mật độ số nang trứng hình thành ở mặt ngoài dạ dày (B) và số thoa trùng ở tuyến nước bọt (D) khi cho các dòng muỗi hút máu có nhiễm giao bào của ký sinh trùng P. falciparum với nồng độ thấp (12)
Những kết quả từ nghiên cứu này là hết sức ấn tượng. Điều đó cho thấy rằng chúng ta có thể sử dụng hệ thống CRISPR/Cas9 không chỉ để làm bất hoạt gene FREP1 mà còn có thể làm bất hoạt những gene khác của loài muỗi mà đóng vai trò quan trọng đối với sự phát triển của ký sinh trùng sốt rét. Đây là tiền đề về công nghệ vô cùng hiệu quả để từ đó chúng ta phát triển các kỹ thuật khác cao hơn trong chiến lược kiểm soát sốt rét, thông qua tạo dòng muỗi biến đổi gene không có khả năng truyền bệnh.
Không dừng lại ở đó, các nhà khoa học của nghiên cứu này có một tham vọng lớn hơn khi muốn trong tương lai có thể thay thế loài muỗi kiểu dại bằng loài muỗi đã được biến đổi gene trong tự nhiên, điều mà trước nay chưa từng được thử nghiệm, mặc dù các nhà khoa học đã và đang phát triển công nghệ “phát động gene” (gene drive). Đây là một công nghệ cho phép lan truyền DNA đã được biến đổi trong quần thể một cách nhanh chóng thông qua giao phối.
Về mặt lý thuyết, việc kết hợp hệ thống CRISPR/Cas9 để tạo dòng muỗi bất hoạt gene FREP1 với công nghệ “phát động gene” (gene drive) để lan truyền sự biến đổi di truyền này trong quần thể kiểu dại ngoài tự nhiên là có thể thực hiện được. Bởi vì việc biến đổi hệ gene của muỗi A. gambiae trong trường hợp này không nhằm vào mục đích làm giảm sức sống hay sức sinh sản của loài, hay nói khác đi “khả năng thích nghi” theo thuyết tiến hóa của Charles Darwin. (15)
Thế nhưng, thực tế lại cho thấy điều ngược lại. Việc gây bất hoạt gene FREP1 mặc dù khiến cho muỗi A. gambiae có khả năng kháng ký sinh trùng sốt rét thế nhưng “khả năng thích nghi” dòng muỗi này lại có phần kém hơn so với dòng muỗi kiểu dại như: khuynh hướng hút máu thấp hơn (blood – feeding propensity), ấu trùng chậm phát triển hơn, vòng đời ngắn hơn, số trứng đẻ của muỗi cái cũng như tỷ lệ trứng nở thấp hơn.
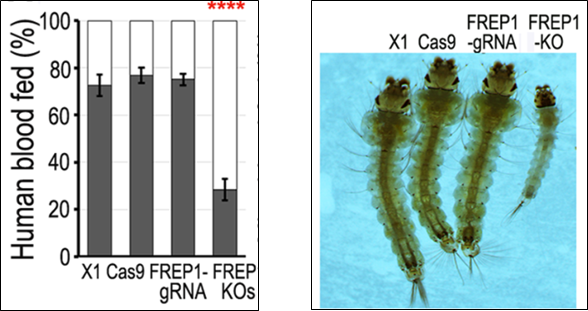
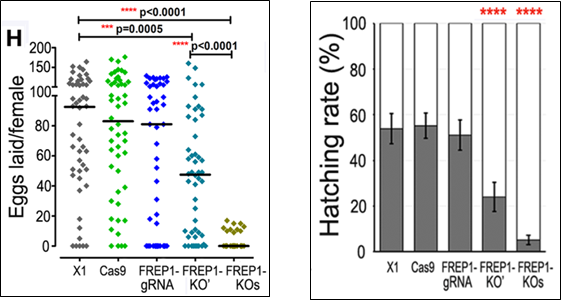
Hình 5. Ảnh hưởng từ việc bất hoạt gene FREP1 đến khả năng thích nghi của các dòng muỗi A. gambiae: (A) khuynh hướng hút máu; (B) ấu trùng chậm phát triển; (C) số trứng đẻ mỗi lứa của muỗi cái; (D) tỷ lệ số trứng nở thành (12)
Sự kém thích nghi này được các nhà khoa học lý giải là bởi FREP1 đóng vai trò quan trọng trong việc hút máu và quá trình tiêu hóa của loài muỗi (14). Vì vậy, việc gây bất hoạt hoàn toàn gene này ở muỗi rất có thể làm giảm khả năng hấp thụ chất dinh dưỡng của muỗi. Sự thiếu hụt dinh dưỡng này khiến cho thời gian phát triển của ấu trùng muỗi bị kéo dài ra hơn so với bình thường. Không những vậy, nó còn làm giảm tỷ lệ đẻ trứng ở muỗi cái.
Một giả thuyết khác được đặt ra đó là hạn chế về mặt công nghệ. Mặc dù hệ thống CRISPR/Cas9 được đánh giá là công cụ biến đổi gene chính xác và hiệu quả thế nhưng hệ thống này trong một số trường hợp vẫn có khả năng gây nên hiệu ứng chệch mục tiêu (off – target effect) ngoài ý muốn mặc dù trong quá trình thao tác, các nhà khoa học đã thiết lập trình tự RNA nhận diện sao cho có tính đặc hiệu cao với gene FREP1 nhằm giảm thiểu đến mức tối đa xảy ra khả năng điều này. Điều đó có nghĩa là trong quá trình hoạt động, bên cạnh việc bất hoạt gene đích FREP1, hệ thống CRISPR/Cas9 rất có thể đã bất hoạt một gene khác tham gia vào quá trình phát triển của muỗi A.gambiae và hậu quả là làm giảm “khả năng thích nghi” của loài muỗi biến đổi gene (16-18).
Để khắc phục những hạn chế của nghiên cứu điều này, các nhà khoa học dự định sẽ tạo ra dòng muỗi A.gambiae chỉ bị bất hoạt gene FREP1 ở dạ dày thay vì toàn bộ. Họ tin rằng việc làm như vậy sẽ giúp giảm thiểu những ảnh hưởng tiêu cực đến “khả năng thích nghi” của loài này.
Thay lời kết, chúng ta sẽ hi vọng rằng trong thời gian tới, các nhà khoa học sẽ đạt được thêm những tiến bộ mới trong nghiên cứu về tạo dòng muỗi có khả năng kháng ký sinh trùng sốt rét. Những kết quả ấy nêu được ứng dụng vào thực tiễn sẽ có thể mang lại những hiệu ứng tích cực trong việc ngăn ngừa dịch bệnh này.
Tài liệu tham khảo:
1. World Health Organization (WHO) (2014) Malaria: fact sheet. (World Health Organization. Regional Office for the Eastern Mediterranean).
2. Center for Disease control and Prevention (CDC) (2018) Biology: Malaria Parasites.
3. Murray CJ, et al. (2012) Global malaria mortality between 1980 and 2010: a systematic analysis. The Lancet 379(9814):413-431.
4. Bùi Đại NVM, Nguyễn Hoàng Tuấn (2005) Bệnh học truyền nhiễm p 231.
5. Nguyễn Trần Chính NHC, Lê Bửu Châu, Nguyễn Văn Hảo, Phan Thị Lệ Hoa, Nguyễn Thế Hùng, Võ Thị Thiên Hương, Cao Ngọc Nga, Nguyễn Duy Phong, Đông Thị Hoài Tâm, Nguyễn Thị Thu Thảo, Đinh Thế Trung (2008) Bệnh truyền nhiễm (Y học) p 397.
6. Ghosh A, Edwards M, & Jacobs-Lorena M (2000) The journey of the malaria parasite in the mosquito: hopes for the new century. Parasitology Today 16(5):196-201.
7. Dinglasan RR, et al. (2007) Disruption of Plasmodium falciparum development by antibodies against a conserved mosquito midgut antigen. Proceedings of the National Academy of Sciences 104(33):13461-13466.
8. Mendes AM, et al. (2008) Conserved mosquito/parasite interactions affect development of Plasmodium falciparum in Africa. PLoS Pathogens 4(5):e1000069.
9. Osta MA, Christophides GK, & Kafatos FC (2004) Effects of mosquito genes on Plasmodium development. science 303(5666):2030-2032.
10. Doudna JA & Charpentier E (2014) The new frontier of genome engineering with CRISPR-Cas9. Science 346(6213):1258096.
11. Hall AB, et al. (2015) A male-determining factor in the mosquito Aedes aegypti. Science 348(6240):1268-1270.
12. Dong Y, Simões ML, Marois E, & Dimopoulos G (2018) CRISPR/Cas9-mediated gene knockout of Anopheles gambiae FREP1 suppresses malaria parasite infection. PLoS pathogens 14(3):e1006898.
13. Li J, et al. (2013) Genome-block expression-assisted association studies discover malaria resistance genes in Anopheles gambiae. Proceedings of the National Academy of Sciences 110(51):20675-20680.
14. Zhang G, et al. (2015) Anopheles midgut FREP1 mediates Plasmodium invasion. Journal of Biological Chemistry:jbc. M114. 623165.
15. John Hopkins Bloomberg School of Public Health (2018) Gene Knockout Using New CRISPR Tool Makes Mosquitoes Highly Resistant to Malaria Parasite.
16. Chen JS, et al. (2017) Enhanced proofreading governs CRISPR–Cas9 targeting accuracy. Nature 550(7676):407.
17. Frock RL, et al. (2015) Genome-wide detection of DNA double-stranded breaks induced by engineered nucleases. Nature biotechnology 33(2):179.
18. Fu Y, et al. (2013) High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nature biotechnology 31(9):822.


